12月9日,中國農業科學院深圳農業基因組研究所阮玨團隊在《自然·方法學(Nature Methods)》上發表第三代測序數據組裝算法wtdbg,該算法極大提高三代測序數據的分析效率,與2019年4月1日發表在《自然·生物技術(Nature Biotechnology)》上的Flye算法相比,分析速度提升了5倍,并首次將測序數據分析時間降低到少于測序數據產出時間。
基因組學技術飛速進步既源于測序技術的發展,同時也依賴于數據分析技術的提高。如今完成一個人的全基因組測序已經是件普通實驗室甚至家庭都可以負擔起費用的“平常”事情,以三代測序為例,完成個人全基因組測序僅需1天時間、費用低于5萬元。但是完成這樣數據規模的全基因組組裝分析,在2014年需要消耗50萬個CPU小時,只能在超大計算機集群上進行。這種情況下,同時對大量個體進行組裝分析是難以想象的,然而以全基因組組裝方式對群體進行測序分析已經成為生物和醫學研究的趨勢。
近年來,生物信息學領域的科學家們致力于改變這種數據產出速度遠高于數據分析速度的尷尬狀況,不斷開發出更高效的組裝分析算法。基因組所在成立之初就布局組學技術研究,致力于將前沿測序技術引入農業科學研究中,阮玨團隊多年來始終專注于測序數據分析方法,如組裝算法的開發,力求推動測序數據的分析速度更快、分析質量更高。
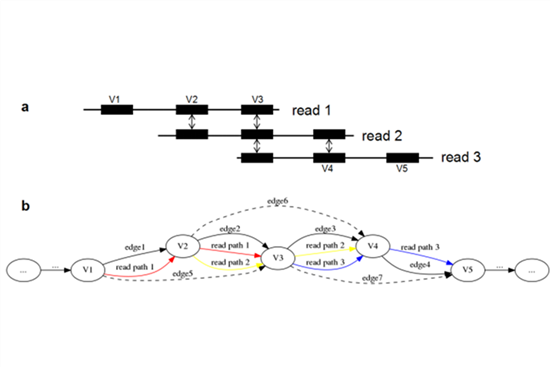
Wtdbg算法的開發得益于一個新的組裝圖理論(模糊布魯因圖)的提出。模糊布魯因圖借鑒了德布魯因圖的思想,將測序數據切分為固定長度的短串,再從短串構建出的圖上恢復出全基因組序列。德布魯因圖以速度優勢常用于第二代測序數據的組裝分析,但因測序噪音極高的難題,從未成功應用在第三代測序數據。模糊布魯因圖重新對短串進行定義,使之能夠容忍高噪音數據,并隨后對生成組裝圖和恢復基因組序列做了大量相應的重構,使其兼具高效率和高容錯的優點。
該項研究始于2013年,由基因組所阮玨研究員與美國哈佛大學醫學院的李恒博士合作完成。研究成果自2016年起對所有人免費開放使用,不僅被幾十篇學術論文引用,還被國內多家基因測序分析公司作為主要組裝分析工具,并且在2019年世界大學生超算競賽中作為性能測試賽題。(通訊員 趙華)
原文鏈接:https://www.nature.com/articles/s41592-019-0669-3